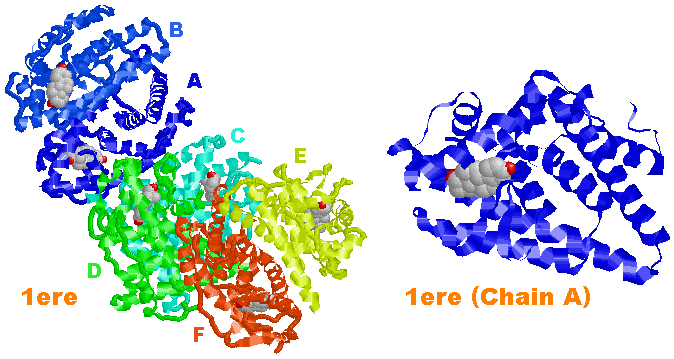
複数のChainを含む場合の特定Chain抽出の例
(1ereの場合;Chain A〜F から A だけをピックアップ)
データのピックアップ
生体分子では,タンパク質やDNAなどが複数分子組み合わされて機能している場合が少なくありません。本サイトで教材としてデータの中には,分子構造をより見やすくするためとデータの読み込み速度を上げるために,その中の一部だけ取り出しているものが多いことをご留意ください。
厳密には全分子を同時に表示する必要があるので,あくまでオリジナルのPDBデータを参照し,そこからリンクされている様々なデータベースの情報を閲覧することをお勧めします。PDB部分データリスト・P450部分データ集からはオリジナルデータへリンクしてあるので,是非ご利用ください。なお,構造データが更新されている場合は,IDの異なる新データが表示されます。
以下には,データの抽出例を示します。

【注】 PDBデータの形式と座標データの抽出
ヘッダ部分には,分類名,データ名,基源生物名,データ作成者名,掲載雑誌名,二次構造情報,SITE情報(PDBデータのLigand結合部位はこれをもとに作成)などが記載されている。
※PDBおよびPDBデータの詳細については,以下をご参照ください。
PDBデータは以下のような情報を記載したテキストデータです。構成原子種とXYZ座標,アミノ酸名(核酸の場合はATGCU)がわかればChimeにより様々な情報を表示できるので,本サイトのデータはその部分だけ抜き出しています。PDBデータのサイズを小さくする秀丸マクロにより,簡便に抜き出しを行うことも可能です。
以下は2fkeのオリジナルデータの抜粋ですが,■で囲んだ部分以外は削除されています。
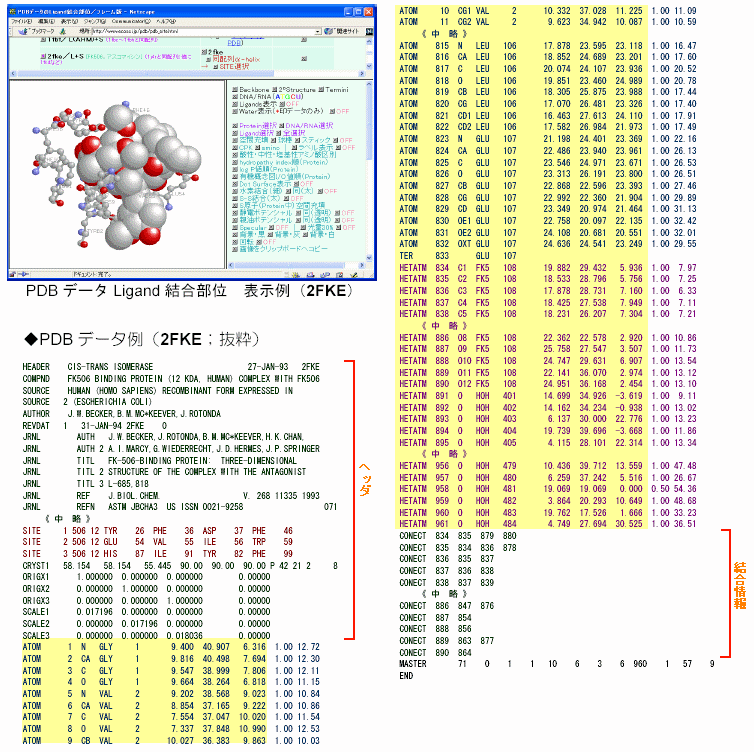
ATOM行が,タンパク質・核酸の構成原子情報で1行の中に以下の内容が書き込まれている。
原子ID,原子種別,残基名(核酸の場合はATGCU),chain ID(A〜;単分子の場合は上のように略記される場合もある),残基番号(核酸の場合は塩基番号),XYZ座標(単位はÅ;1Å=10-10m),占有率,温度因子
HETATM行には,タンパク質・核酸以外の分子(リガンド,水など)の原子種,XYZ座標などが記載されている。