- The Crystal Structure of a Sodium Galactose Transporter Reveals Mechanistic Insights into Na+/Sugar Symport(Science,2008/08/08)
- ヒトの60兆個の細胞にまつわるそれぞれのドラマ。細胞の奥深い営みを読み解く---永田和宏・著『細胞の不思議』(現代ビジネス,2015/04/12)
- 永田和宏さんほかによる研究:小胞体レドックスタンパク質ERp18の亜鉛イオン依存的な機能制御機構を解明(京都産業大学,2024/02/08) → アルツハイマー病治療薬(本サイト)
- Na+/グルコース共輸送体 - meddic | シンポート
- ナトリウム-グルコース共輸送タンパクの発見 - Wikipedia
- 元素・分子が登場する本を読もう!(本サイト) | 別トピック(本サイト;グルコース輸送体)
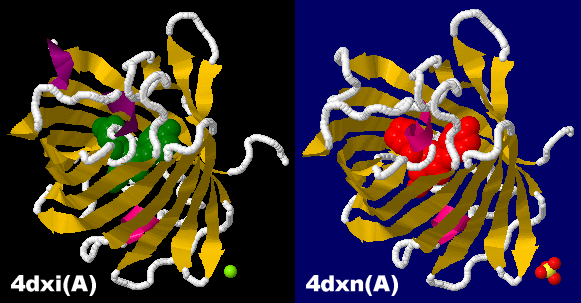
PDBデータ 3dh4のChain A(ガラクトースが結合したNa+/糖共輸送体)
[Glucose transporter - Wikipedia,Symporter - Wikipedia]
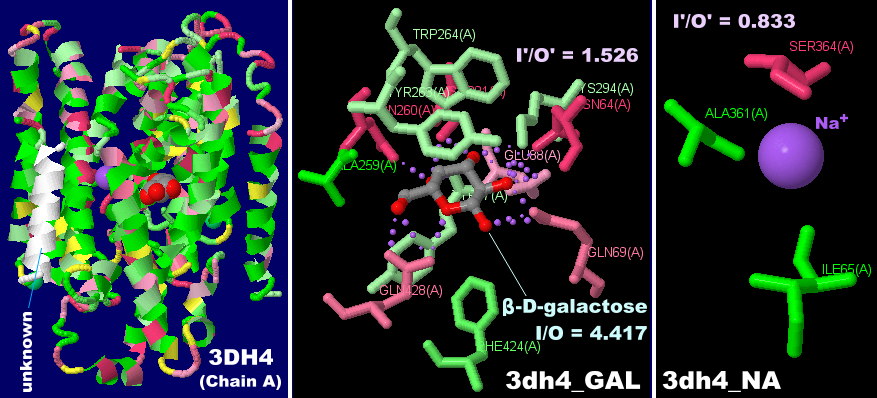
β-D-ガラクトースとNa+が結合したNa+/糖共輸送体3dh4のChain AとPDBsumデータ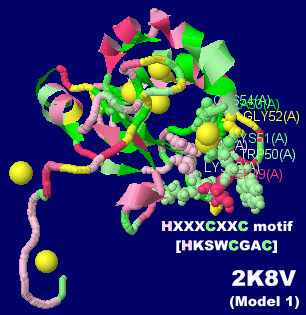
ERp18の構造例2k8vのModel 1(アミノ酸残基47-54のHKSWCGACを球表示,黄色空間充填はシステイン中のS原子)
| 1-40 | Met | His | His | His | His | His | His | Met | Ser | Asp | Gly | His | Asn | Gly | Leu | Gly | Lys | Gly | Phe | Gly | Asp | His | Ile | His | Trp | Arg | Thr | Leu | Glu | Asp | Gly | Lys | Lys | Glu | Ala | Ala | Ala | Ser | Gly | Leu |
| 41-80 | Pro | Leu | Met | Val | Ile | Ile | His | Lys | Ser | Trp | Cys | Gly | Ala | Cys | Lys | Ala | Leu | Lys | Pro | Lys | Phe | Ala | Glu | Ser | Thr | Glu | Ile | Ser | Glu | Leu | Ser | His | Asn | Phe | Val | Met | Val | Asn | Leu | Glu |
| 81-120 | Asp | Glu | Glu | Glu | Pro | Lys | Asp | Glu | Asp | Phe | Ser | Pro | Asp | Gly | Gly | Tyr | Ile | Pro | Arg | Ile | Leu | Phe | Leu | Asp | Pro | Ser | Gly | Lys | Val | His | Pro | Glu | Ile | Ile | Asn | Glu | Asn | Gly | Asn | Pro |
| 121-157 | Ser | Tyr | Lys | Tyr | Phe | Tyr | Val | Ser | Ala | Glu | Gln | Val | Val | Gln | Gly | Met | Lys | Glu | Ala | Gln | Glu | Arg | Leu | Thr | Gly | Asp | Ala | Phe | Arg | Lys | Lys | His | Leu | Glu | Asp | Glu | Leu |
-
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
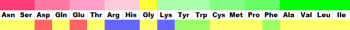
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
42残基のアミロイドβのアミノ酸配列
2mscのChain Aのアミノ酸配列(1行50残基;I/O値順着色)
MERS-CoVおよびSARS-CoVのプロテアーゼのアミノ酸配列比較(何れもChain A,I/O値順着色)
[Ebola virus disease - Wikipedia,Nucleoprotein - Wikipedia]
[Amyloid beta - Wikipedia]
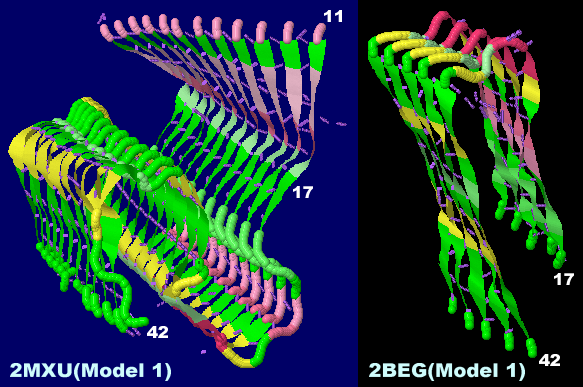
2mxuと2begの構造比較(何れもModel 1,数字は残基番号)
※FASTA形式: DAEFRHDSGYEVHHQKLVFFAEDVGSNKGAIIGLMVGGVVIA → 自作秀丸マクロで下表作成
残基番号
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
残基
Asp
Ala
Glu
Phe
Arg
His
Asp
Ser
Gly
Tyr
Glu
Val
His
His
Gln
Lys
Leu
Val
Phe
Phe
Ala
Glu
Asp
Val
Gly
Ser
Asn
Lys
Gly
Ala
Ile
Ile
Gly
Leu
Met
Val
Gly
Gly
Val
Val
Ile
Ala
[Dopamine transporter - Wikipedia]
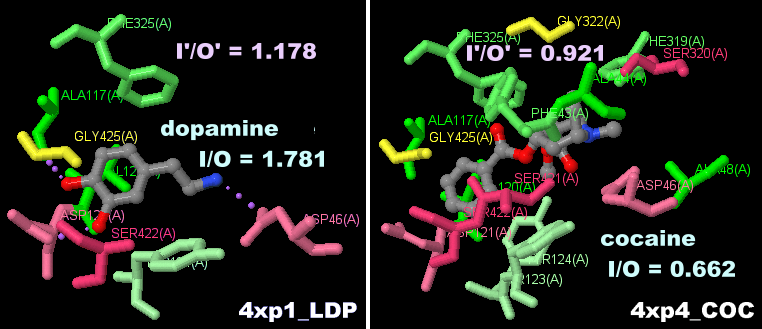
ドーパミンが結合したキイロショウジョウバエのドーパミン輸送体4xp1のChain A(左)およびPDBsumデータ(右;コカイン結合の4xp4と比較)
※RCSB PDBのFacebook記事(2015/05/22)参照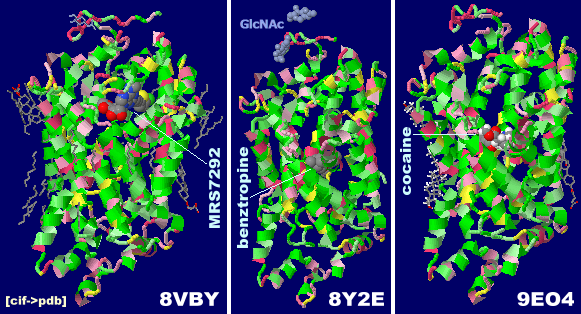
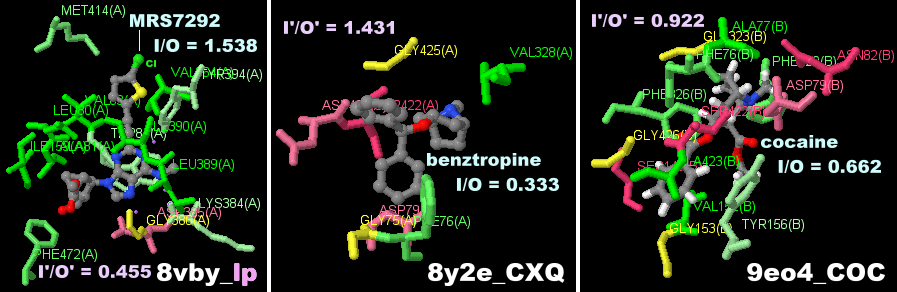
[上]ヒトのドーパミン(ドパミン)輸送体 左から 8vby(MRS7292結合),8y2e(ベンズトロピン結合),9eo4(コカイン結合)
[下]それぞれのPDBsumデータ
[Barrier-to-autointegration factor - Wikipedia]
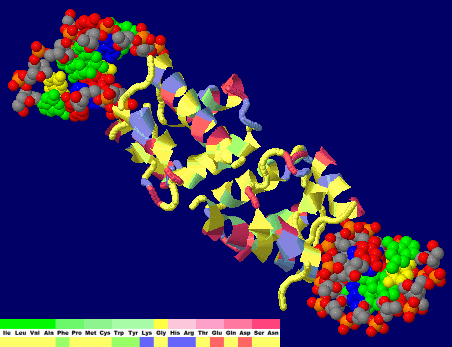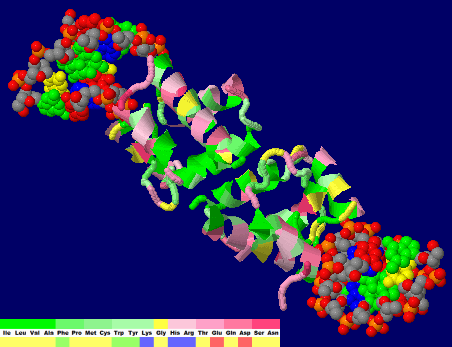
DNAとBAFの結合構造例2bzfの6量体(Chain A-C×2)
[Lenvatinib - Wikipedia,VEGF receptors - Wikipedia]
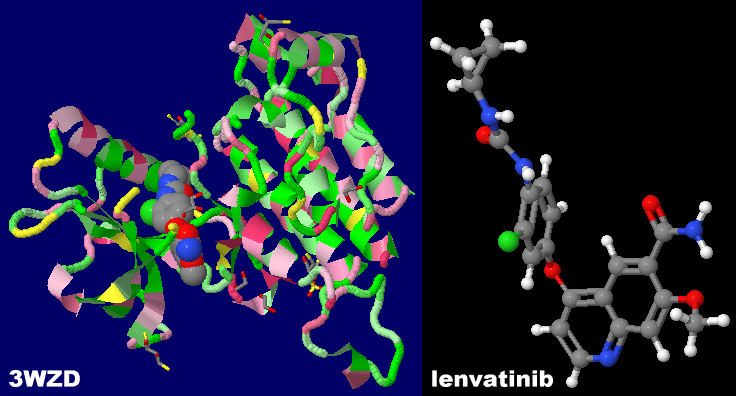
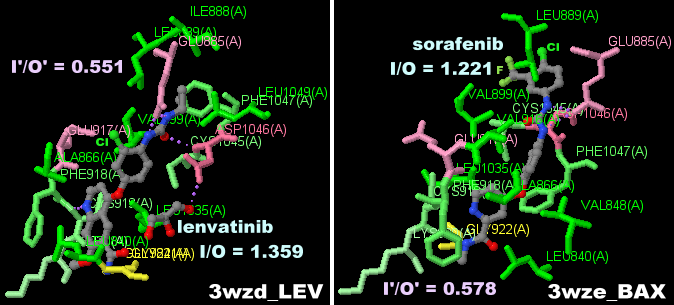
[左]血管内皮増殖因子レセプター2 3wzdとレンバチニブ [右]3wzd(レンバチニブ〈lenvatinib〉結合)と3wze(ソラフェニブ〈sorafenib〉結合)のPDBsumデータ
[RAGE (receptor) - Wikipedia,Glycated hemoglobin - Wikipedia]
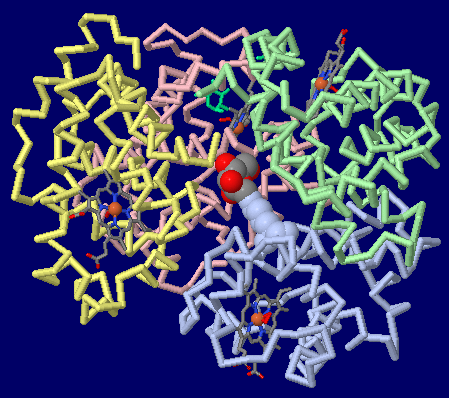
糖化ヘモグロビン3b75のChain A-D(空間充填表示はChain Aのフルクトースとその結合アミノ酸残基Lys 99)
[KRAS - Wikipedia,Nanodisc - Wikipedia]
Gly
Pro
Leu
Lys
Leu
Leu
Asp
Asn
Trp
Asp
Ser
Val
Thr
Ser
Thr
Phe
Ser
Lys
Leu
Arg
Glu
Gln
Leu
Gly
Pro
Val
Thr
Gln
Glu
Phe
Trp
Asp
Asn
Leu
Glu
Lys
Glu
Thr
Glu
Gly
Leu
Arg
Gln
Glu
Met
Ser
Lys
Asp
Leu
Glu
Glu
Val
Lys
Ala
Lys
Val
Gln
Pro
Tyr
Leu
Asp
Asp
Phe
Gln
Lys
Lys
Trp
Gln
Glu
Glu
Met
Glu
Leu
Tyr
Arg
Gln
Lys
Val
Glu
Pro
Leu
Arg
Ala
Glu
Leu
Gln
Glu
Gly
Ala
Arg
Gln
Lys
Leu
His
Glu
Leu
Gln
Glu
Lys
Leu
Ser
Pro
Leu
Gly
Glu
Glu
Met
Arg
Asp
Arg
Ala
Arg
Ala
His
Val
Asp
Ala
Leu
Arg
Thr
His
Leu
Ala
Pro
Tyr
Ser
Asp
Glu
Leu
Arg
Gln
Arg
Leu
Ala
Ala
Arg
Leu
Glu
Ala
Leu
Lys
Glu
Asn
Gly
Gly
Ala
Arg
Leu
Ala
Glu
Tyr
His
Ala
Lys
Ala
Thr
Glu
His
Leu
Ser
Thr
Leu
Ser
Glu
Lys
Ala
Lys
Pro
Ala
Leu
Glu
Asp
Leu
Arg
Gln
Gly
Leu
Leu
Pro
Val
Leu
Glu
Ser
Phe
Lys
Val
Ser
Phe
Leu
Ser
Ala
Leu
Glu
Glu
Tyr
Thr
Lys
Lys
Leu
Asn
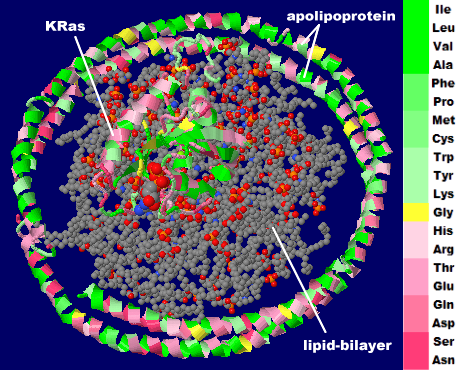
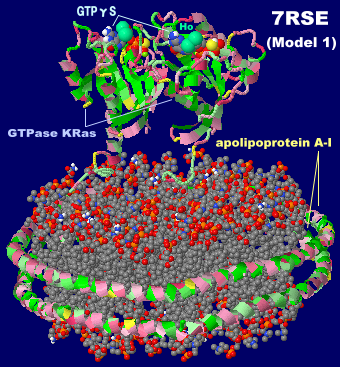
脂質二重膜ナノディスクに結合したGTPアーゼKRas2mscのModel 1(左)と同7rseのModel 1(右)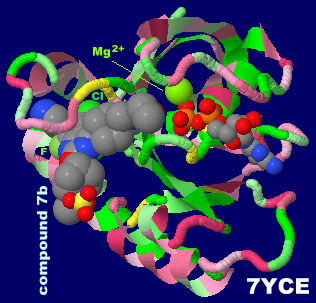
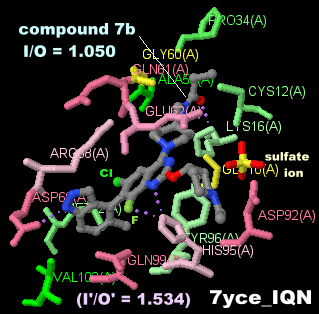
阻害剤compound 7bが結合したKRas G12C変異体7yceとPDBsumデータ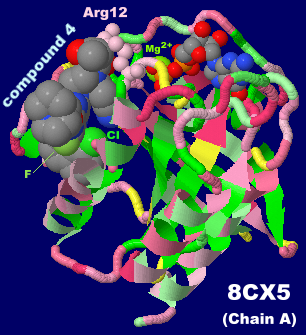
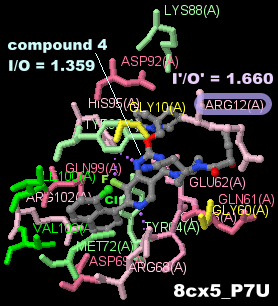
阻害剤compound 4(α,β-ketoamide 4)が結合したKRas G12C変異体8cx5のChain AとPDBsumデータ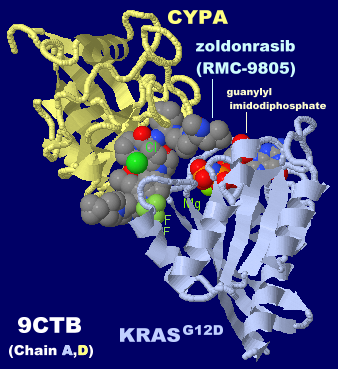
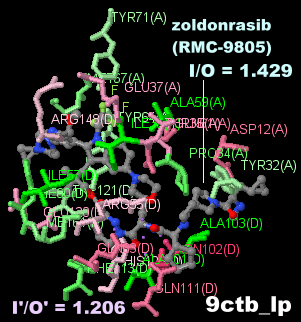
阻害剤zoldonrasib(RMC-9805)が結合したKRas G12D変異体とCYPA複合体9ctbのChain A・DとPDBsumデータ[204(A)]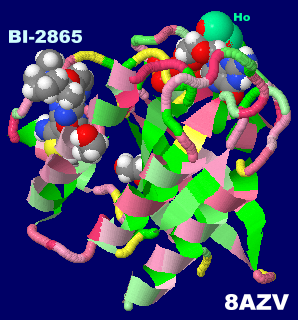
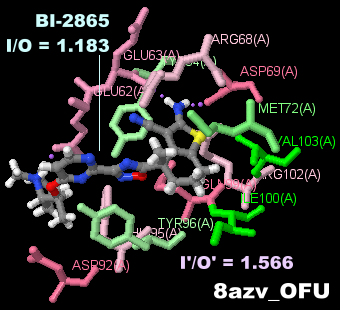
[左]阻害剤BI-2865が結合したPan-Kras 8azvとPDBsumデータ
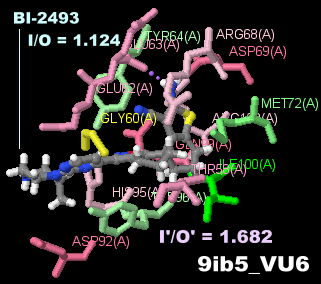
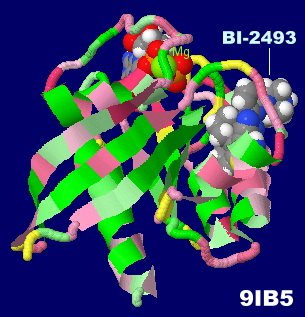
[右]阻害剤BI-2493が結合したPan-Kras 9ib5とPDBsumデータ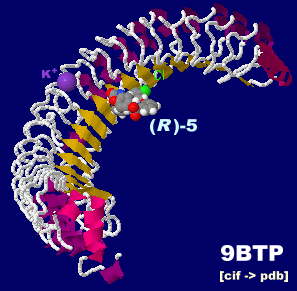
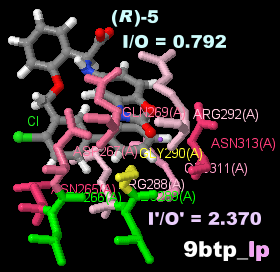
阻害剤(R)-5が結合したSHOC2 9btpとPDBsumデータ
[Middle East respiratory syndrome coronavirus - Wikipedia]
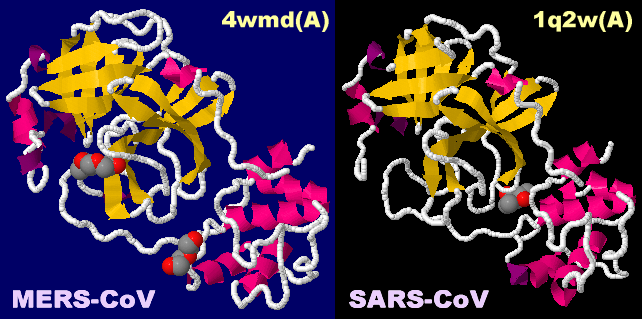
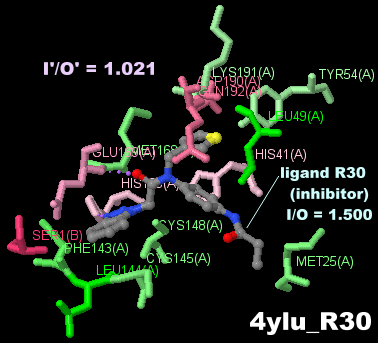
[左・中]MERS-CoVのプロテアーゼ4wmdのChain AとSARS-CoVのプロテアーゼ1q2wのChain A [右]阻害剤が結合したMERS-CoVのプロテアーゼ4yluのPDBsumデータ
※Chain Aについては,4wmd・4wme・4wmfのアミノ酸配列,4rsp・4yluのアミノ酸配列は同一。両者は赤字の148番残基のみ異なる。
MERS(4wmd)1-50
Ser
Gly
Leu
Val
Lys
Met
Ser
His
Pro
Ser
Gly
Asp
Val
Glu
Ala
Cys
Met
Val
Gln
Val
Thr
Cys
Gly
Ser
Met
Thr
Leu
Asn
Gly
Leu
Trp
Leu
Asp
Asn
Thr
Val
Trp
Cys
Pro
Arg
His
Val
Met
Cys
Pro
Ala
Asp
Gln
Leu
Ser
MERS(4rsp)1-50
Ser
Gly
Leu
Val
Lys
Met
Ser
His
Pro
Ser
Gly
Asp
Val
Glu
Ala
Cys
Met
Val
Gln
Val
Thr
Cys
Gly
Ser
Met
Thr
Leu
Asn
Gly
Leu
Trp
Leu
Asp
Asn
Thr
Val
Trp
Cys
Pro
Arg
His
Val
Met
Cys
Pro
Ala
Asp
Gln
Leu
Ser
SARS(1q2w)1-50
Ser
Leu
Ser
Gly
Phe
Arg
Lys
Met
Ala
Phe
Pro
Ser
Gly
Lys
Val
Glu
Gly
Cys
Met
Val
Gln
Val
Thr
Cys
Gly
Thr
Thr
Thr
Leu
Asn
Gly
Leu
Trp
Leu
Asp
Asp
Thr
Val
Tyr
Cys
Pro
Arg
His
Val
Ile
Cys
Thr
Ala
Glu
Asp
MERS(4wmd)51-100
Asp
Pro
Asn
Tyr
Asp
Ala
Leu
Leu
Ile
Ser
Met
Thr
Asn
His
Ser
Phe
Ser
Val
Gln
Lys
His
Ile
Gly
Ala
Pro
Ala
Asn
Leu
Arg
Val
Val
Gly
His
Ala
Met
Gln
Gly
Thr
Leu
Leu
Lys
Leu
Thr
Val
Asp
Val
Ala
Asn
Pro
Ser
MERS(4rsp)51-100
Asp
Pro
Asn
Tyr
Asp
Ala
Leu
Leu
Ile
Ser
Met
Thr
Asn
His
Ser
Phe
Ser
Val
Gln
Lys
His
Ile
Gly
Ala
Pro
Ala
Asn
Leu
Arg
Val
Val
Gly
His
Ala
Met
Gln
Gly
Thr
Leu
Leu
Lys
Leu
Thr
Val
Asp
Val
Ala
Asn
Pro
Ser
SARS(1q2w)51-100
Met
Leu
Asn
Pro
Asn
Tyr
Glu
Asp
Leu
Leu
Ile
Arg
Lys
Ser
Asn
His
Ser
Phe
Leu
Val
Gln
Ala
Gly
Asn
Val
Gln
Leu
Arg
Val
Ile
Gly
His
Ser
Met
Gln
Asn
Cys
Leu
Leu
Arg
Leu
Lys
Val
Asp
Thr
Ser
Asn
Pro
Lys
Thr
MERS(4wmd)101-150
Thr
Pro
Ala
Tyr
Thr
Phe
Thr
Thr
Val
Lys
Pro
Gly
Ala
Ala
Phe
Ser
Val
Leu
Ala
Cys
Tyr
Asn
Gly
Arg
Pro
Thr
Gly
Thr
Phe
Thr
Val
Val
Met
Arg
Pro
Asn
Tyr
Thr
Ile
Lys
Gly
Ser
Phe
Leu
Cys
Gly
Ser
Ala
Gly
Ser
MERS(4rsp)101-150
Thr
Pro
Ala
Tyr
Thr
Phe
Thr
Thr
Val
Lys
Pro
Gly
Ala
Ala
Phe
Ser
Val
Leu
Ala
Cys
Tyr
Asn
Gly
Arg
Pro
Thr
Gly
Thr
Phe
Thr
Val
Val
Met
Arg
Pro
Asn
Tyr
Thr
Ile
Lys
Gly
Ser
Phe
Leu
Cys
Gly
Ser
Cys
Gly
Ser
SARS(1q2w)101-150
Pro
Lys
Tyr
Lys
Phe
Val
Arg
Ile
Gln
Pro
Gly
Gln
Thr
Phe
Ser
Val
Leu
Ala
Cys
Tyr
Asn
Gly
Ser
Pro
Ser
Gly
Val
Tyr
Gln
Cys
Ala
Met
Arg
Pro
Asn
His
Thr
Ile
Lys
Gly
Ser
Phe
Leu
Asn
Gly
Ser
Cys
Gly
Ser
Val
MERS(4wmd)151-200
Val
Gly
Tyr
Thr
Lys
Glu
Gly
Ser
Val
Ile
Asn
Phe
Cys
Tyr
Met
His
Gln
Met
Glu
Leu
Ala
Asn
Gly
Thr
His
Thr
Gly
Ser
Ala
Phe
Asp
Gly
Thr
Met
Tyr
Gly
Ala
Phe
Met
Asp
Lys
Gln
Val
His
Gln
Val
Gln
Leu
Thr
Asp
MERS(4rsp)151-200
Val
Gly
Tyr
Thr
Lys
Glu
Gly
Ser
Val
Ile
Asn
Phe
Cys
Tyr
Met
His
Gln
Met
Glu
Leu
Ala
Asn
Gly
Thr
His
Thr
Gly
Ser
Ala
Phe
Asp
Gly
Thr
Met
Tyr
Gly
Ala
Phe
Met
Asp
Lys
Gln
Val
His
Gln
Val
Gln
Leu
Thr
Asp
SARS(1q2w)151-200
Gly
Phe
Asn
Ile
Asp
Tyr
Asp
Cys
Val
Ser
Phe
Cys
Tyr
Met
His
His
Met
Glu
Leu
Pro
Thr
Gly
Val
His
Ala
Gly
Thr
Asp
Leu
Glu
Gly
Lys
Phe
Tyr
Gly
Pro
Phe
Val
Asp
Arg
Gln
Thr
Ala
Gln
Ala
Ala
Gly
Thr
Asp
Thr
MERS(4wmd)201-250
Lys
Tyr
Cys
Ser
Val
Asn
Val
Val
Ala
Trp
Leu
Tyr
Ala
Ala
Ile
Leu
Asn
Gly
Cys
Ala
Trp
Phe
Val
Lys
Pro
Asn
Arg
Thr
Ser
Val
Val
Ser
Phe
Asn
Glu
Trp
Ala
Leu
Ala
Asn
Gln
Phe
Thr
Glu
Phe
Val
Gly
Thr
Gln
Ser
MERS(4rsp)201-250
Lys
Tyr
Cys
Ser
Val
Asn
Val
Val
Ala
Trp
Leu
Tyr
Ala
Ala
Ile
Leu
Asn
Gly
Cys
Ala
Trp
Phe
Val
Lys
Pro
Asn
Arg
Thr
Ser
Val
Val
Ser
Phe
Asn
Glu
Trp
Ala
Leu
Ala
Asn
Gln
Phe
Thr
Glu
Phe
Val
Gly
Thr
Gln
Ser
SARS(1q2w)201-250
Ile
Thr
Leu
Asn
Val
Leu
Ala
Trp
Leu
Tyr
Ala
Ala
Val
Ile
Asn
Gly
Asp
Arg
Trp
Phe
Leu
Asn
Arg
Phe
Thr
Thr
Thr
Leu
Asn
Asp
Phe
Asn
Leu
Val
Ala
Met
Lys
Tyr
Asn
Tyr
Glu
Pro
Leu
Thr
Gln
Asp
His
Val
Asp
Ile
MERS(4wmd)251-300
Val
Asp
Met
Leu
Ala
Val
Lys
Thr
Gly
Val
Ala
Ile
Glu
Gln
Leu
Leu
Tyr
Ala
Ile
Gln
Gln
Leu
Tyr
Thr
Gly
Phe
Gln
Gly
Lys
Gln
Ile
Leu
Gly
Ser
Thr
Met
Leu
Glu
Asp
Glu
Phe
Thr
Pro
Glu
Asp
Val
Asn
Met
Gln
Ile
MERS(4rsp)251-300
Val
Asp
Met
Leu
Ala
Val
Lys
Thr
Gly
Val
Ala
Ile
Glu
Gln
Leu
Leu
Tyr
Ala
Ile
Gln
Gln
Leu
Tyr
Thr
Gly
Phe
Gln
Gly
Lys
Gln
Ile
Leu
Gly
Ser
Thr
Met
Leu
Glu
Asp
Glu
Phe
Thr
Pro
Glu
Asp
Val
Asn
Met
Gln
Ile
SARS(1q2w)251-300
Leu
Gly
Pro
Leu
Ser
Ala
Gln
Thr
Gly
Ile
Ala
Val
Leu
Asp
Met
Cys
Ala
Ala
Leu
Lys
Glu
Leu
Leu
Gln
Asn
Gly
Met
Asn
Gly
Arg
Thr
Ile
Leu
Gly
Ser
Thr
Ile
Leu
Glu
Asp
Glu
Phe
Thr
Pro
Phe
Asp
Val
Val
Arg
Gln
MERS(4wmd)301-
Met
Gly
Val
Val
Met
Gln
MERS(4rsp)301-
Met
Gly
Val
Val
Met
Gln
SARS(1q2w)301-
Cys
Ser
Gly
Val
Thr
Glu
Gly