[Deoxyhypusine synthase - Wikipedia]
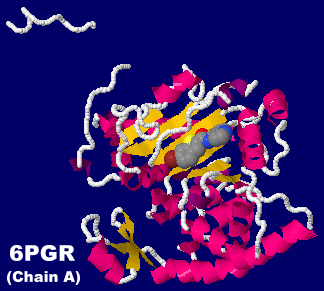
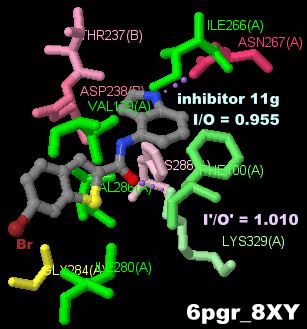
阻害剤が結合したデオキシハイプシン合成酵素6pgrのChain AとそのPDBsumデータ
-
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
[Lipase - Wikipedia,Orlistat - Wikipedia]
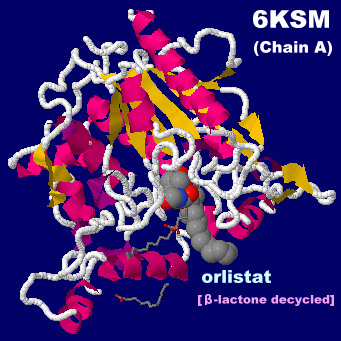
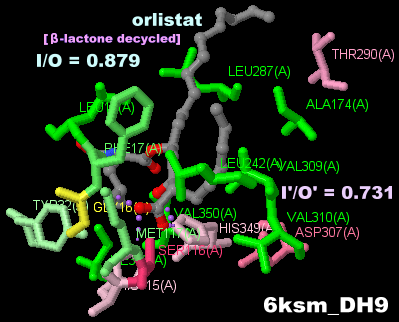
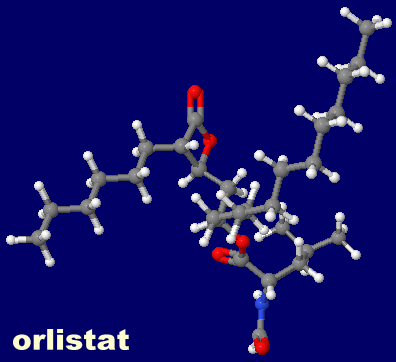
[左・中]阻害剤オルリスタット(β-ラクトン開環体)が結合したリパーゼ6ksmのChain AとそのPDBsumデータ [右]オルリスタット(orlistat)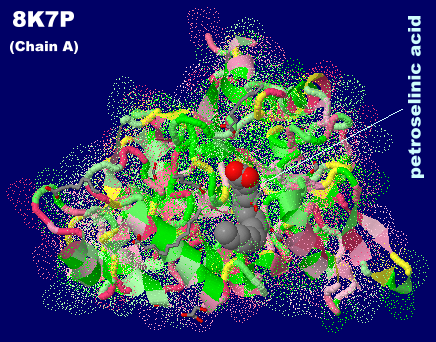
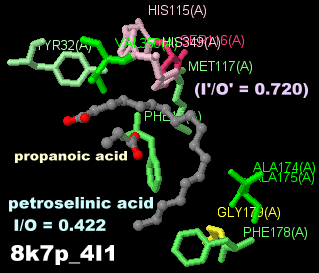
ペトロセリン酸(petroselinic acid)が結合したリパーゼ 8k7pのChain AとそのPDBsumデータ
[Porphyromonas gingivalis - Wikipedia]

ジンジバリス菌(歯周病 病原細菌)線毛のFimAピリンの重合体6kmf
[ATP-binding cassette transporter - Wikipedia]
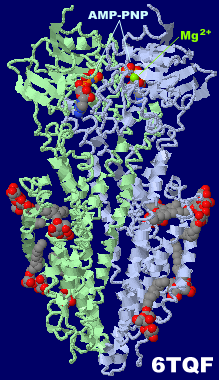
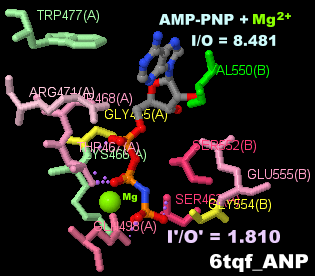
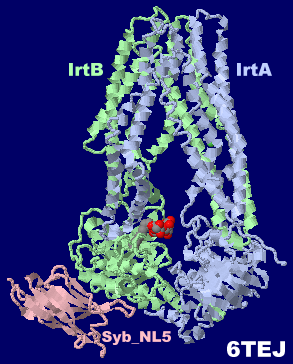
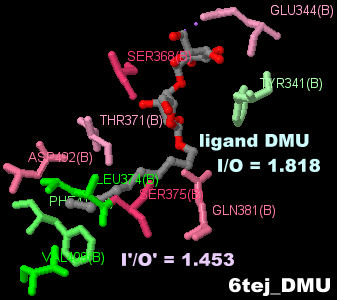
[左]AMP-PNPが結合したABC輸送体Rv1819c 6tqfとそのPDBsumデータ [右]Syb_NL5が結合したABC輸送体IrtAB 6tejとそのPDBsumデータ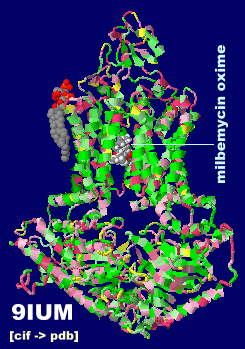
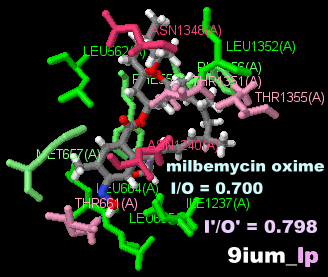
ミルベマイシン・オキシム(milbemycin oxime)が結合したABC輸送体Cdr1 9iumとPDBsumデータ
[PETase - Wikipedia]
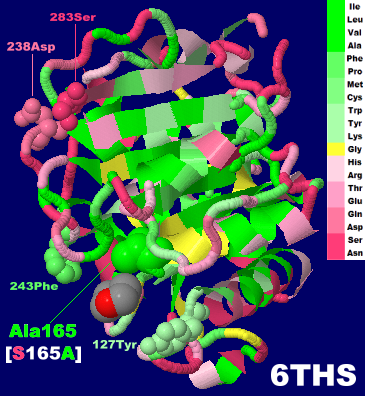
PETヒドロラーゼ(S165A変異体)6ths
[Amine oxidase (copper-containing) - Wikipedia,Topaquinone - Wikipedia]
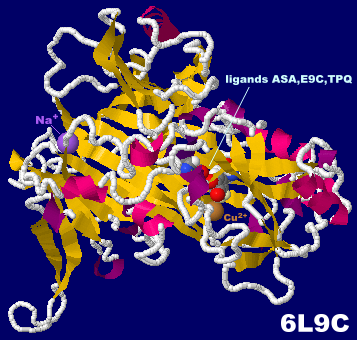
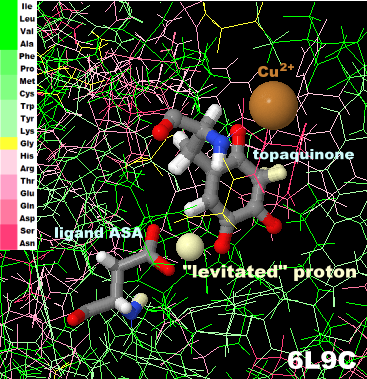
銅アミン酸化酵素6l9cとその"宙に浮いた"プロトン周辺の拡大図
[Polyketide synthase - Wikipedia]
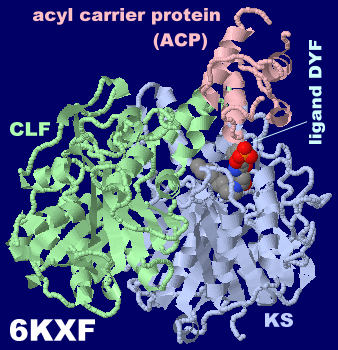
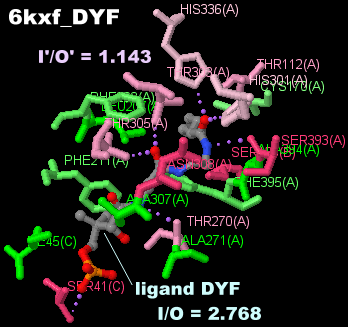
II型ポリケタイド(ポリケチド)合成酵素6kxfとそのPDBsumデータ
[Gibberellin - Wikipedia]
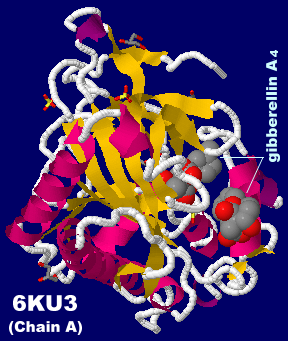
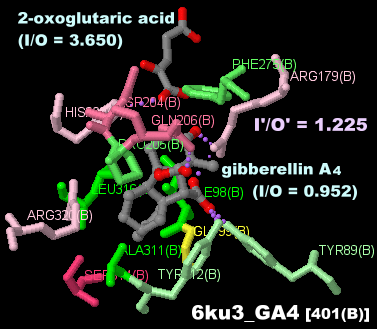
[左]ジベレリンA4が結合したジベレリン2-オキシダーゼ3(GA2ox3)6ku3のChain AとそのPDBsumデータ(こちらはChain Bから)
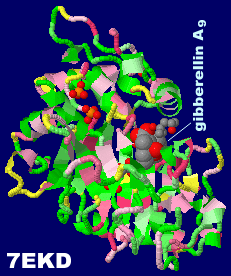
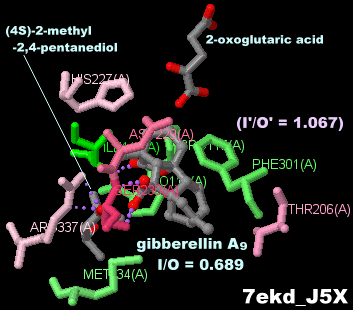
[右]同7ekd(ジベレリンA9結合)とそのPDBsumデータ
[DGAT1 - Wikipedia]
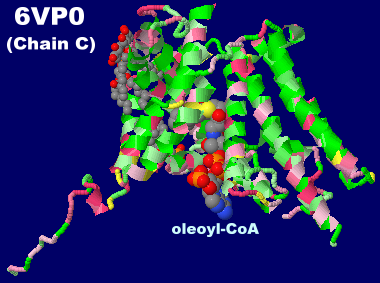
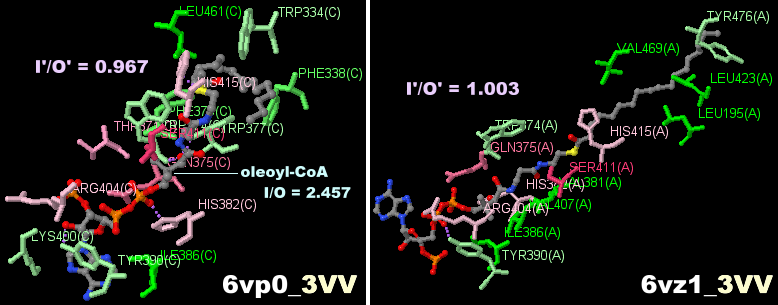
オレオイル-CoAが結合したジアシルグリセロールO-アシルトランスフェラーゼ1(DGAT1)6vp0のChain CとそのPDBsumデータ(別研究の6vz1との比較)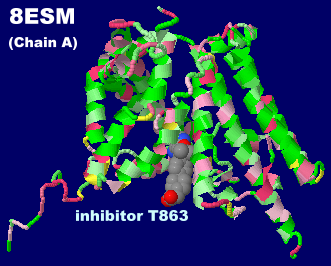
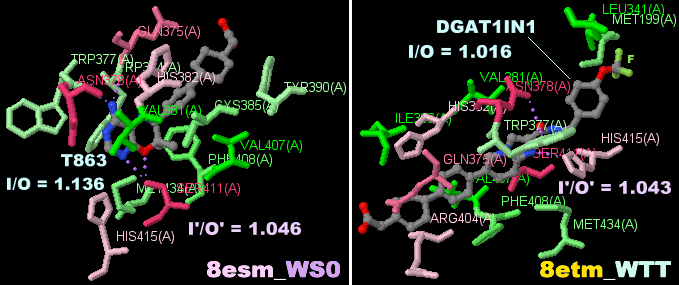
阻害剤T863が結合したDGAT1 8esmのChain AとそのPDBsumデータ(阻害剤DGAT1IN1結合の8etmとの比較)
[ACAT1 - Wikipedia]
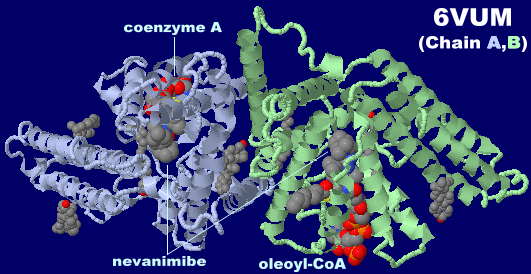
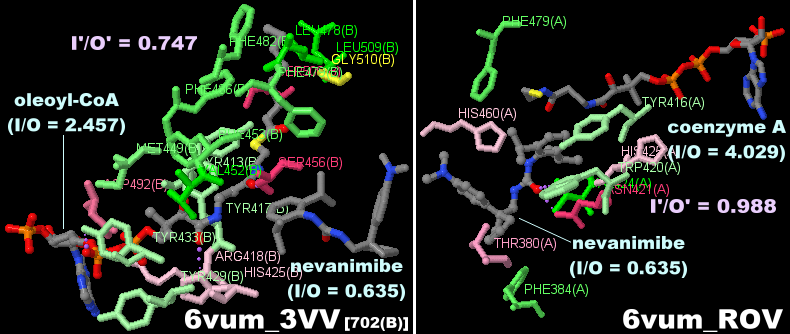
オレオイル-CoA,CoA,阻害剤nevanimibeなどが結合したアシルCoA:コレステロールO-アシルトランスフェラーゼ1(ACAT1)6vumのChain A・BとそのPDBsumデータ