- Evolution of abiotic cubane chemistries in a nucleic acid aptamer allows selective recognition of a malaria biomarker(PNAS,2020/07/06)
- @PDBeuropeによる関連ツイート
- マラリアとは - 厚生労働省-戸山研究庁舎
- 今月の分子 102: 乳酸脱水素酵素(Lactate Dehydrogenase)(PDBj,2008/06) | Molecule of the Month(PDB)
- 乳酸脱水素酵素の別研究
- 乳酸脱水素酵素 - Wikipedia | マラリア
[Lactate dehydrogenase - Wikipedia,Plasmodium vivax - Wikipedia]
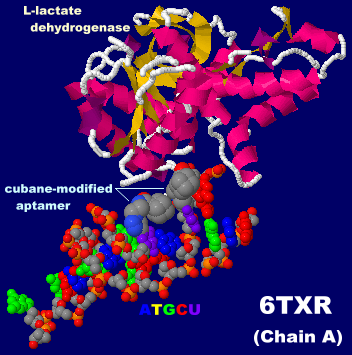
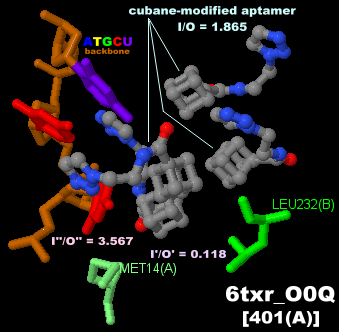
キュバン修飾型アプタマーが結合した三日熱マラリア原虫の乳酸脱水素酵素(PvLDH)6txrのChain AとPDBsumデータ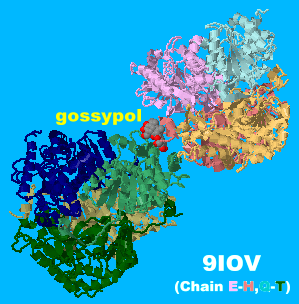
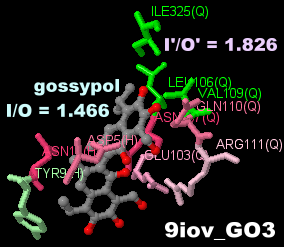
ゴシポール(gossypol)が結合したヒトの乳酸脱水素酵素A 9iovのChain E-H・Q-TとPDBsumデータ
-
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
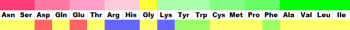
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
フィトスルホカイン受容体1のアミノ酸配列(4z64のChain A)
[CALHM1 - Wikipedia]
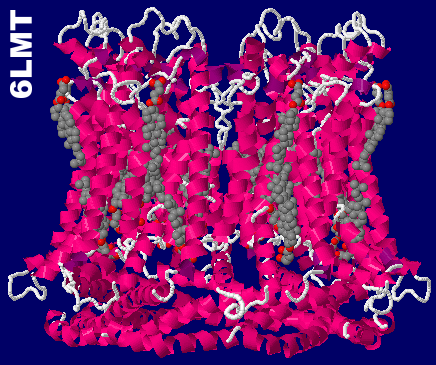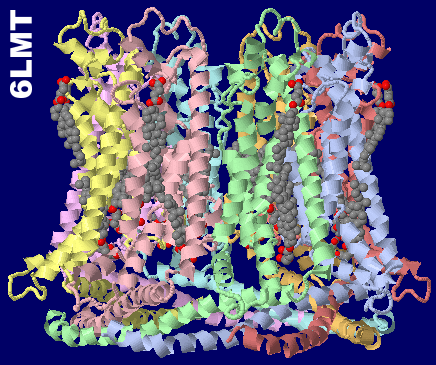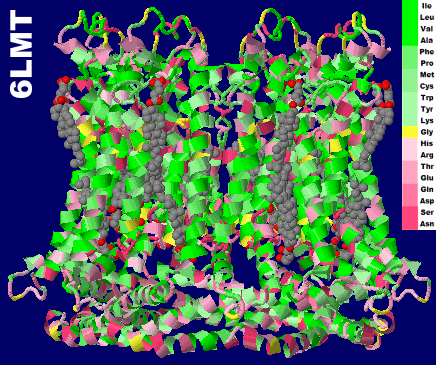
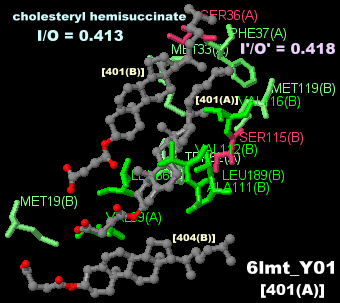
CALHM1(カルシウム恒常性モジュレータ1)6lmtとそのPDBsumデータ(ヘミコハク酸コレステリル,[401(A)])

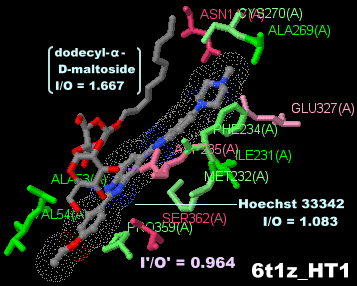
多剤トランスポーターLmrP 6t1zとそのPDBsumデータ
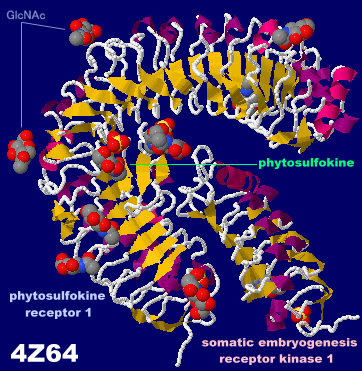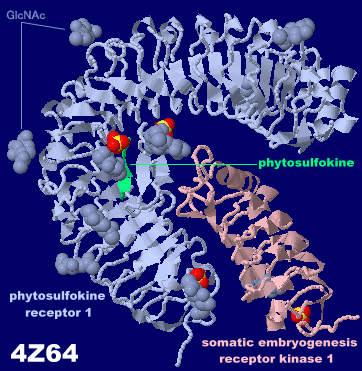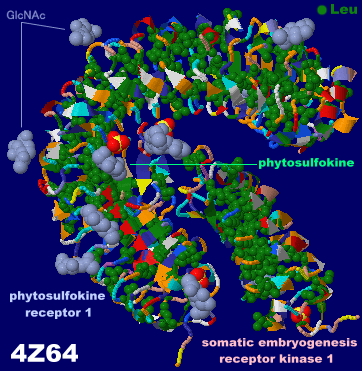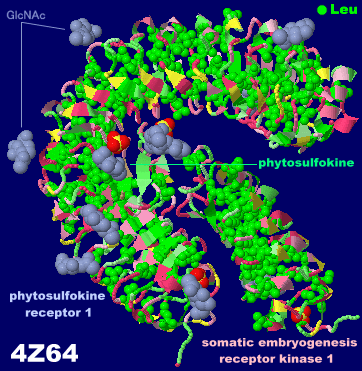
フィトスルホカイン(phytosulfokine)補助受容体タンパク質SERK1が結合したフィトスルホカイン受容体1 4z64
Leu = 100 (15.8 %)
Glu
Ser
Gln
Thr
Thr
Ser
Arg
Cys
His
Pro
His
Asp
Leu
Glu
Ala
Leu
Arg
Asp
Phe
Ile
Ala
His
Leu
Glu
Pro
Lys
Pro
Asp
Gly
Trp
Ile
Asn
Ser
Ser
Ser
Ser
Thr
Asp
Cys
Cys
Asn
Trp
Thr
Gly
Ile
Thr
Cys
Asn
Ser
Asn
Asn
Thr
Gly
Arg
Val
Ile
Arg
Leu
Glu
Leu
Gly
Asn
Lys
Lys
Leu
Ser
Gly
Lys
Leu
Ser
Glu
Ser
Leu
Gly
Lys
Leu
Asp
Glu
Ile
Arg
Val
Leu
Asn
Leu
Ser
Arg
Asn
Phe
Ile
Lys
Asp
Ser
Ile
Pro
Leu
Ser
Ile
Phe
Asn
Leu
Lys
Asn
Leu
Gln
Thr
Leu
Asp
Leu
Ser
Ser
Asn
Asp
Leu
Ser
Gly
Gly
Ile
Pro
Thr
Ser
Ile
Asn
Leu
Pro
Ala
Leu
Gln
Ser
Phe
Asp
Leu
Ser
Ser
Asn
Lys
Phe
Asn
Gly
Ser
Leu
Pro
Ser
His
Ile
Cys
His
Asn
Ser
Thr
Gln
Ile
Arg
Val
Val
Lys
Leu
Ala
Val
Asn
Tyr
Phe
Ala
Gly
Asn
Phe
Thr
Ser
Gly
Phe
Gly
Lys
Cys
Val
Leu
Leu
Glu
His
Leu
Cys
Leu
Gly
Met
Asn
Asp
Leu
Thr
Gly
Asn
Ile
Pro
Glu
Asp
Leu
Phe
His
Leu
Lys
Arg
Leu
Asn
Leu
Leu
Gly
Ile
Gln
Glu
Asn
Arg
Leu
Ser
Gly
Ser
Leu
Ser
Arg
Glu
Ile
Arg
Asn
Leu
Ser
Ser
Leu
Val
Arg
Leu
Asp
Val
Ser
Trp
Asn
Leu
Phe
Ser
Gly
Glu
Ile
Pro
Asp
Val
Phe
Asp
Glu
Leu
Pro
Gln
Leu
Lys
Phe
Phe
Leu
Gly
Gln
Thr
Asn
Gly
Phe
Ile
Gly
Gly
Ile
Pro
Lys
Ser
Leu
Ala
Asn
Ser
Pro
Ser
Leu
Asn
Leu
Leu
Asn
Leu
Arg
Asn
Asn
Ser
Leu
Ser
Gly
Arg
Leu
Met
Leu
Asn
Cys
Thr
Ala
Met
Ile
Ala
Leu
Asn
Ser
Leu
Asp
Leu
Gly
Thr
Asn
Arg
Phe
Asn
Gly
Arg
Leu
Pro
Glu
Asn
Leu
Pro
Asp
Cys
Lys
Arg
Leu
Lys
Asn
Val
Asn
Leu
Ala
Arg
Asn
Thr
Phe
His
Gly
Gln
Val
Pro
Glu
Ser
Phe
Lys
Asn
Phe
Glu
Ser
Leu
Ser
Tyr
Phe
Ser
Leu
Ser
Asn
Ser
Ser
Leu
Ala
Asn
Ile
Ser
Ser
Ala
Leu
Gly
Ile
Leu
Gln
His
Cys
Lys
Asn
Leu
Thr
Thr
Leu
Val
Leu
Thr
Leu
Asn
Phe
His
Gly
Glu
Ala
Leu
Pro
Asp
Asp
Ser
Ser
Leu
His
Phe
Glu
Lys
Leu
Lys
Val
Leu
Val
Val
Ala
Asn
Cys
Arg
Leu
Thr
Gly
Ser
Met
Pro
Arg
Trp
Leu
Ser
Ser
Ser
Asn
Glu
Leu
Gln
Leu
Leu
Asp
Leu
Ser
Trp
Asn
Arg
Leu
Thr
Gly
Ala
Ile
Pro
Ser
Trp
Ile
Gly
Asp
Phe
Lys
Ala
Leu
Phe
Tyr
Leu
Asp
Leu
Ser
Asn
Asn
Ser
Phe
Thr
Gly
Glu
Ile
Pro
Lys
Ser
Leu
Thr
Lys
Leu
Glu
Ser
Leu
Thr
Ser
Arg
Asn
Ile
Ser
Val
Asn
Glu
Pro
Ser
Pro
Asp
Phe
Pro
Phe
Phe
Met
Lys
Arg
Asn
Glu
Ser
Ala
Arg
Ala
Leu
Gln
Tyr
Asn
Gln
Ile
Phe
Gly
Phe
Pro
Pro
Thr
Ile
Glu
Leu
Gly
His
Asn
Asn
Leu
Ser
Gly
Pro
Ile
Trp
Glu
Glu
Phe
Gly
Asn
Leu
Lys
Lys
Leu
His
Val
Phe
Asp
Leu
Lys
Trp
Asn
Ala
Leu
Ser
Gly
Ser
Ile
Pro
Ser
Ser
Leu
Ser
Gly
Met
Thr
Ser
Leu
Glu
Ala
Leu
Asp
Leu
Ser
Asn
Asn
Arg
Leu
Ser
Gly
Ser
Ile
Pro
Val
Ser
Leu
Gln
Gln
Leu
Ser
Phe
Leu
Ser
Lys
Phe
Ser
Val
Ala
Tyr
Asn
Asn
Leu
Ser
Gly
Val
Ile
Pro
Ser
Gly
Gly
Gln
Phe
Gln
Thr
Phe
Pro
Asn
Ser
Ser
Phe
Glu
Ser
Asn
His
Leu
Cys
Gly
Glu
His
Arg
Phe
Pro
Cys
Ser
Glu
Gly
Thr
Glu
Ser
Ala
Leu
Ile
Lys
His
His
His
His
His
His
Ile = 32 (5.1 %)
Val = 20 (3.2 %)
Cys = 13 (2.1 %)
Total = 631
[5α-Reductase - Wikipedia,Finasteride - Wikipedia]
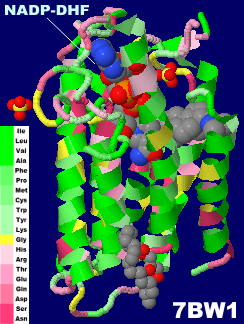
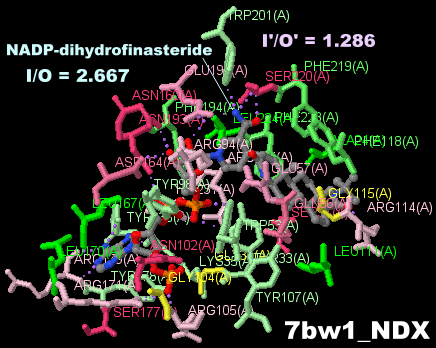
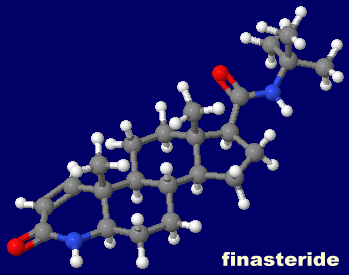
[左・中]NADP-ジヒドロフィナステリドが結合した3-オキソ-5α-ステロイド-4-デヒドロゲナーゼ2 7bw1とそのPDBsumデータ [右]フィナステリド(finasteride)
[Ribonuclease P - Wikipedia,Pentatricopeptide repeat - Wikipedia]
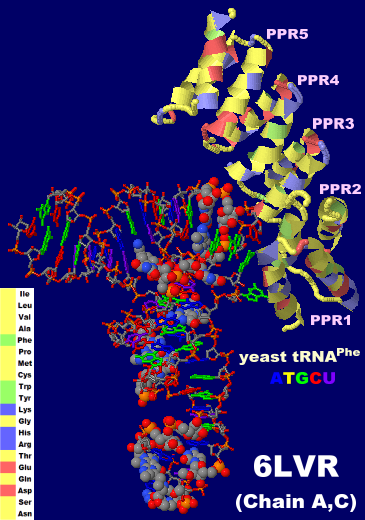
エンザイム型リボヌクレアーゼPのPPR(ペンタトリコペプチドリピート)ドメインとtRNA複合体立体構造 6lvrのChain A・C
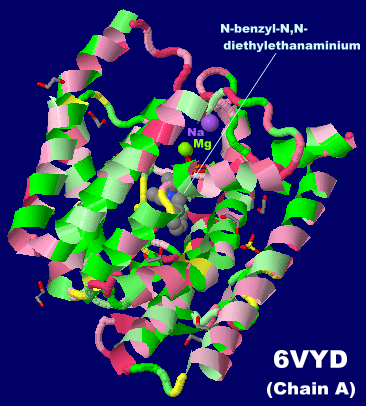
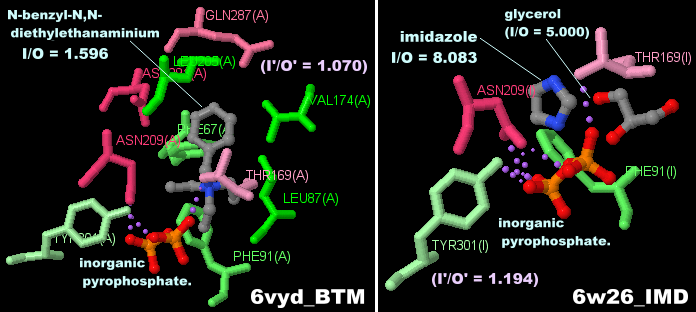
N-ベンジル-N,Nジエチルエタンアミニウムが結合したテルペン環化酵素 FgGS 6vydのChain AとそのPDBsumデータ(イミダゾール結合の6w26との比較)
[Trypsin 1 - Wikipedia,5-Methoxytryptamine - Wikipedia]
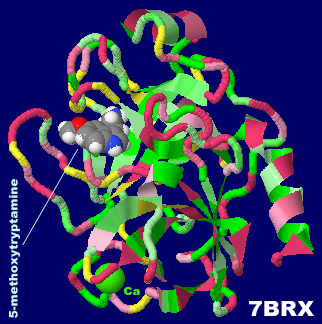
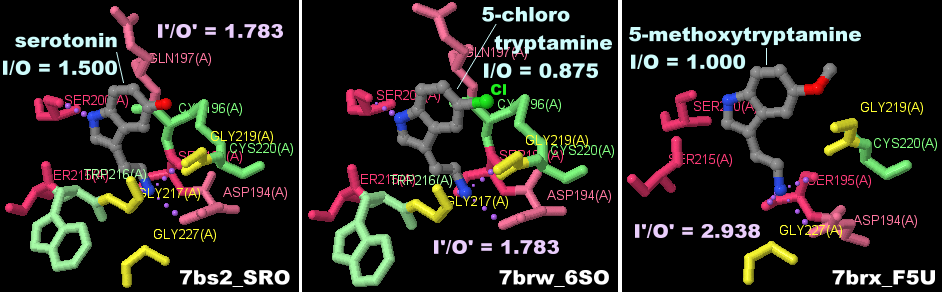
[左]5-メトキシトリプタミンが結合したウシ膵臓トリプシン7brx
[右]膵臓トリプシンのPDBsumデータ比較:7bs2(セロトニン),7brw(5-クロロトリプタミン),7brx(5-メトキシトリプタミン)
[HIV - Wikipedia,HIV/AIDS - Wikipedia,Lenacapavir - Wikipedia]

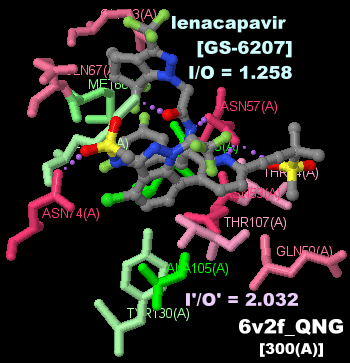
レナカパビル(lenacapavir,GS-6207;カプシド阻害剤)が結合したHIV-1カプシド 6v2fのChain AとそのPDBsumデータ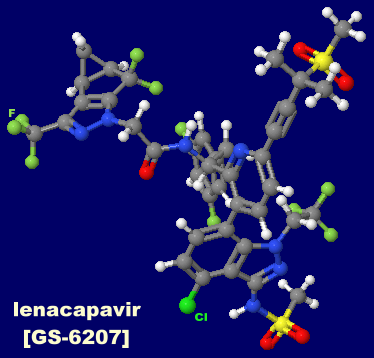
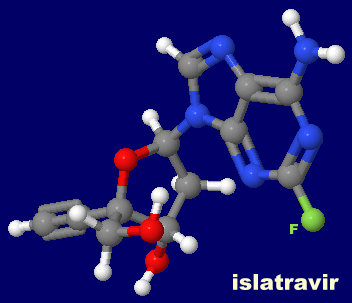
[左]レナカパビル(lenacapavir,GS-6207;カプシド阻害剤) [右]イスラトラビル(islatravir;核酸系逆転写酵素阻害剤)
[Pannexin - Wikipedia,PANX1 - Wikipedia,Carbenoxolone - Wikipedia]
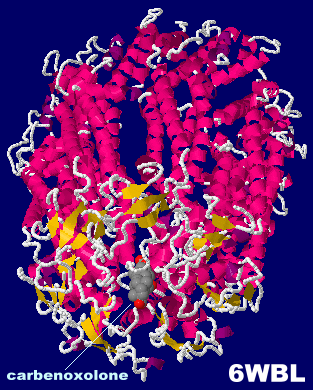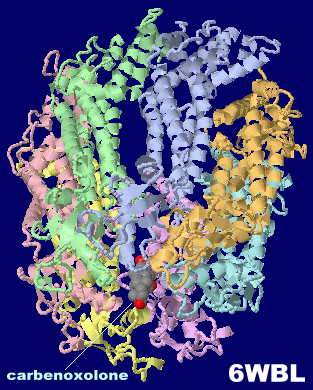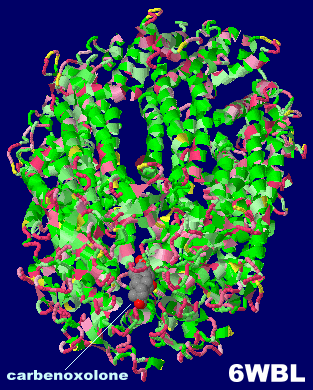
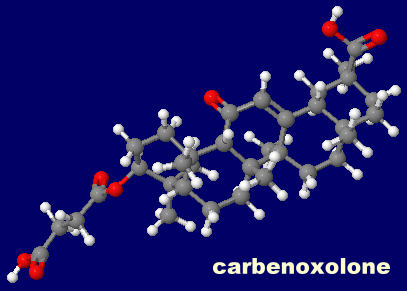
カルベノキソロン(carbenoxolone)が結合したヒトパネキシン1 6wbl(PDBsumデータに近傍アミノ酸無し;最近は各ChainのTrp)とカルベノキソロン