- Maturation of the functional mouse CRES amyloid from globular form(PNAS,2020/06/29) ※下掲6uio
- 医療用医薬品 : ポビドンヨード - KEGG ※ポビドンヨードとアミロースとしてオブラートを使えばヨウ素デンプン反応実験可能
- 参考(ヨウ素を取り上げた番組例):がっちりマンデー!!「儲かる元素 原子番号53 I ヨウ素」(2009/02/15放送)
- 甲状腺ホルモンと受容体(本サイト) | 代表的な高分子
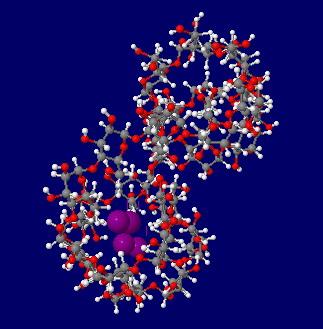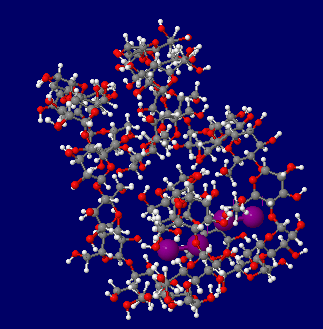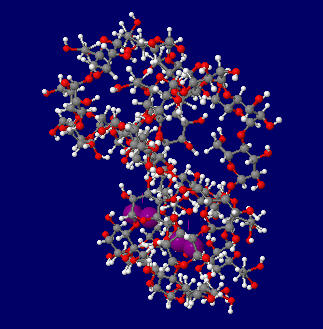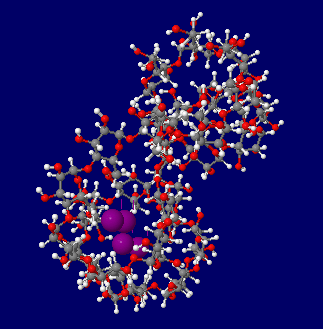
ヨウ素デンプン反応模式図:
26量体シクロアミロース構造1c58にI2を2分子添加(厳密な分子計算はしていない)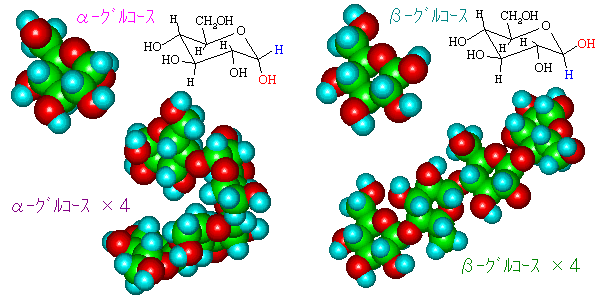
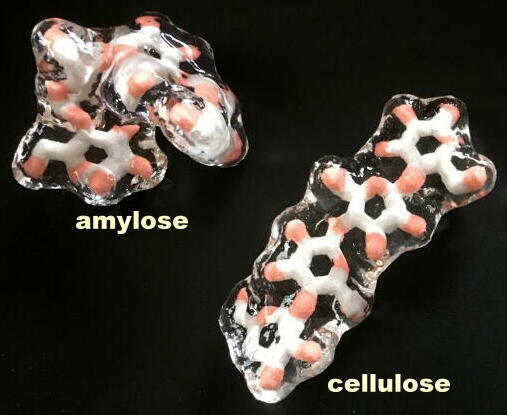
ヨウ素デンプン反応におけるコイル構造のアミロース(α-グルコース重合体の部分構造;β-グルコース重合体セルロースの部分構造と並べて)
※右写真はその川上モデル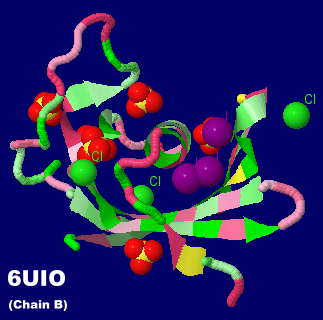
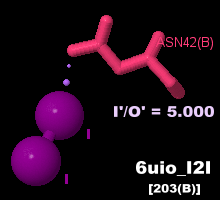
ヨウ素分子結合タンパク質例:6uioのChain B(シスタチン関連タンパク質CRES)とPDBsumデータ
-
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
●ヒトの必須・非必須区別
ILE LEU VAL PHE MET TRP LYS HIS THR ARG ALA PRO CYS TYR GLY GLU GLN ASP SER ASN
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
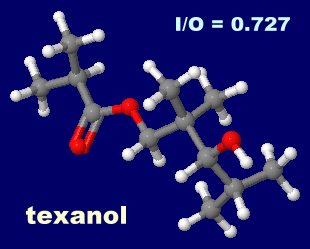
texanol(テキサノール)
I = 345, O = 465, I/O = 0.742
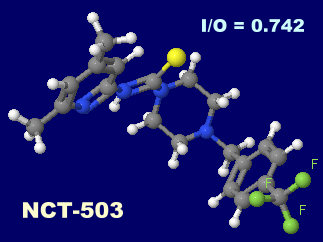
NCT-503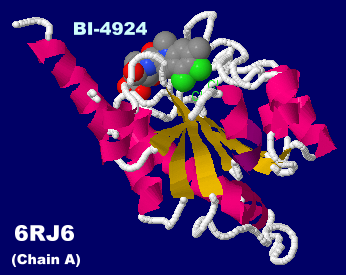
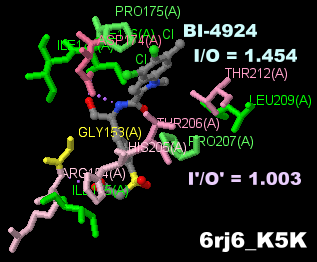
阻害剤BI-4924が結合したホスホグリセリン酸デヒドロゲナーゼ(PHGDH)6rj6のChain AとPDBsumデータ
※Journal of Medicinal Chemistry誌論文(2019/07/31)参照

表紙画像(関連書籍と並べて)
I = 160, O = 220, I/O = 0.727
p.7:
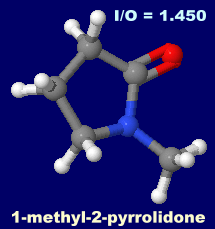
[左]1-methyl-2-pyrrolidone(1-メチル-2-ピロリドン)
[右]texanol(テキサノール)
p.85:ヨウ素デンプン反応模式図(26量体シクロアミロース構造1c58にI2を2分子添加;厳密な分子計算はしていない)
→ 別トピック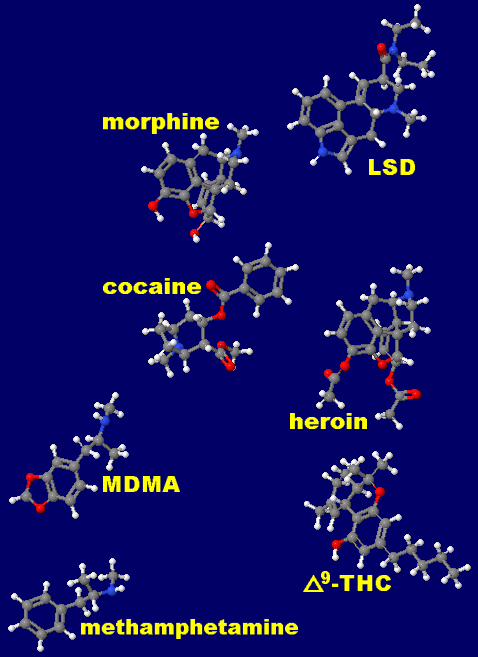

p.173:[左]morphine(モルヒネ)ほかドラッグ例の分子モデル
[右]ドラッグ例分子群の有機概念図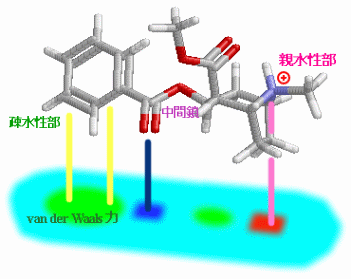
p.173:cocaine(コカイン)の受容体との結合模式図
※山川ほか「メディシナルケミストリー 第4版」(講談社)pp.125-126
→ コカインを含む生体分子のSITE例
[Deubiquitinating enzyme - Wikipedia]
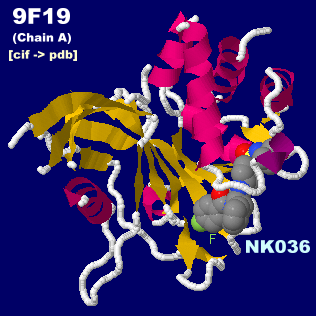
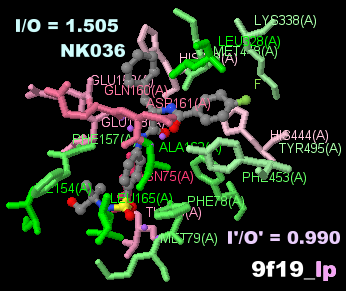
阻害剤NK036が結合したユビキチン特異的プロテアーゼUSP30キメラ9f19のChain AとPDBsumデータ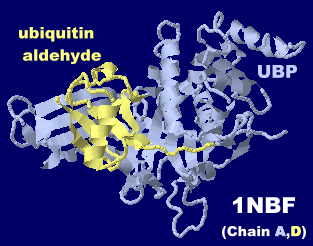
参考:ユビキチンアルデヒドが結合したユビキチン特異的プロテアーゼ(UBP)1nbfのChain A,D
→ 分子モデルで見るノーベル賞(本サイト;2004年ノーベル化学賞/ユビキチン)
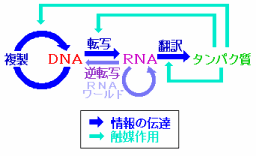 セントラルドグマの模式図
セントラルドグマの模式図
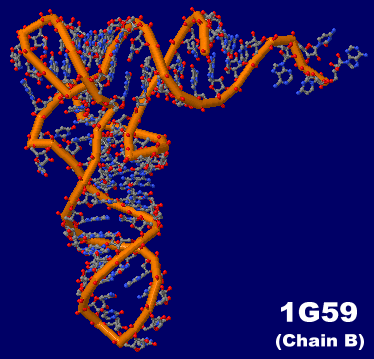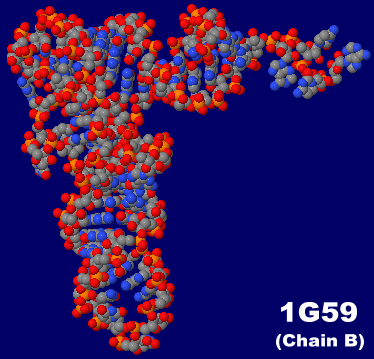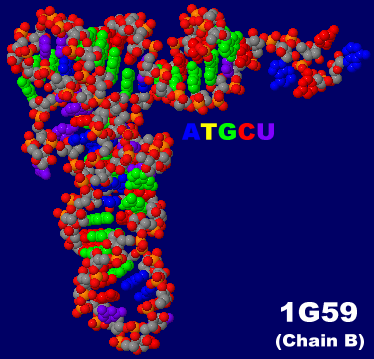
[左]p.3ほか多数 転移RNA(tRNA)例1g59(tRNAGlu)のChain B
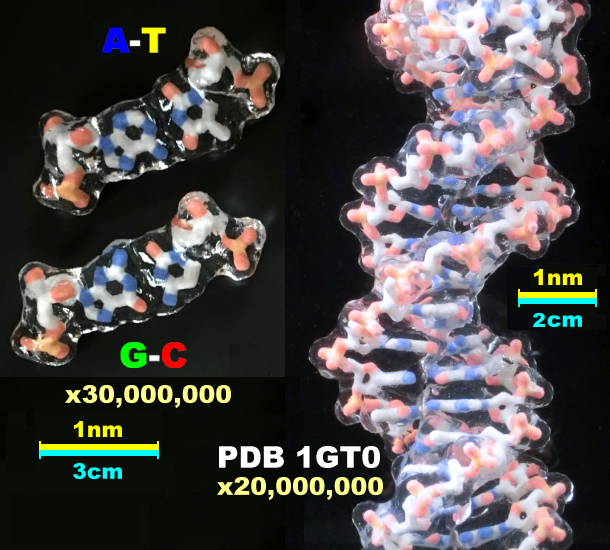
[右]p.11ほか多数 DNA例1gt0の3Dプリンタ模型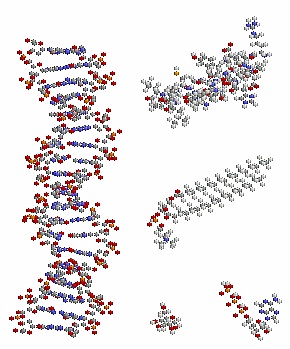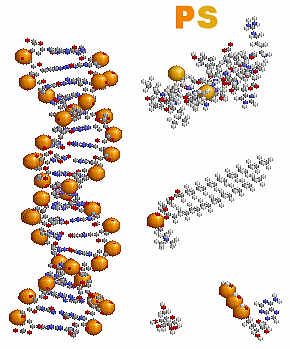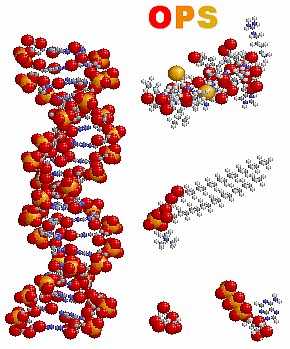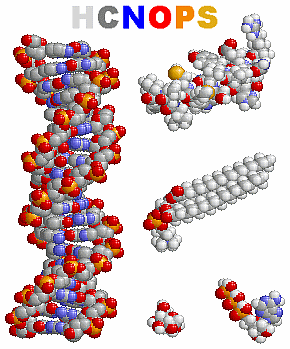
p.76 生化学物質の大部分はこの6種類の元素だけからできている(C・N・H・O・P・S):
→ 生体分子の構成元素
I = 1279, O = 820, I/O = 1.560
[Colibactin - Wikipedia]
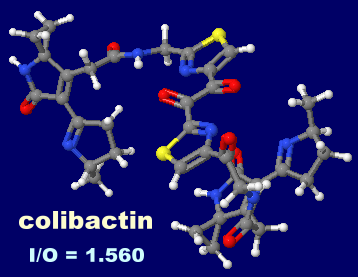
コリバクチン(colibactin)
※参考:ピロリ菌による発がん関連(2005年 ノーベル生理学医学賞関連)
[左]ピロリ菌がんタンパク質CagA 4dvy [右]ピロリ菌の好中球活性化タンパク質1ji4
[Anthocyanidin reductase - Wikipedia]
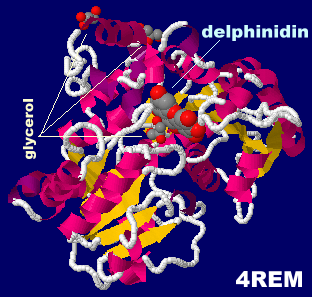
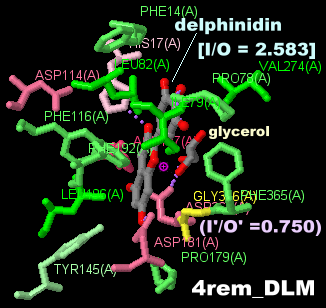
デルフィニジン(delphinidin)が結合したアントシアニジン3-O-グルコシルトランスフェラーゼ4remとPDBsumデータ(-O+= は -OH で計算)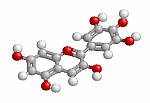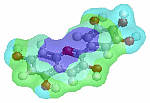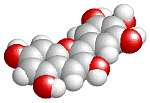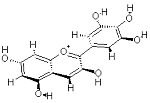
デルフィニジン(平板構造のdelphinidin;旧Chime版で画像作成)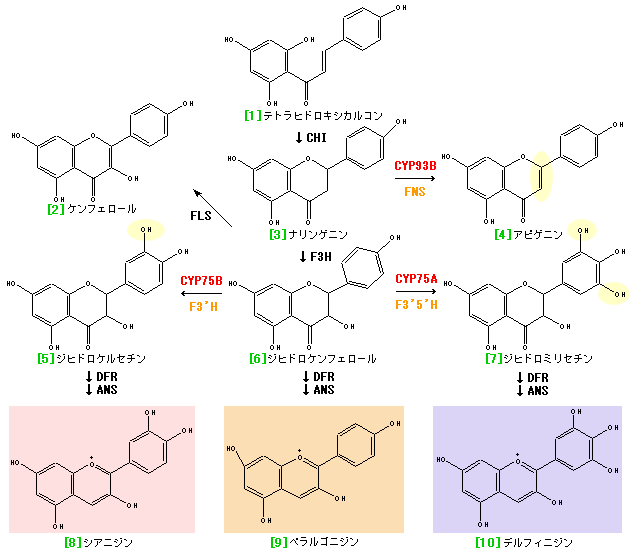
フラボノイドの生合成経路(抜粋) → フラボノイドの生合成経路
※大村恒雄・石村巽・藤井義明 編,「P450の分子生物学 第2版」,講談社サイエンティフィク(2009)
I = 299, O = 350, I/O = 0.854
[6PPD - Wikipedia]
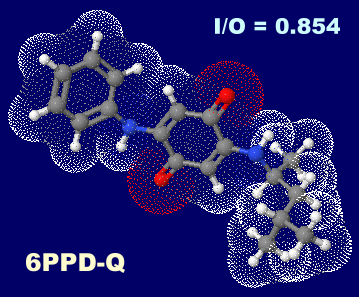
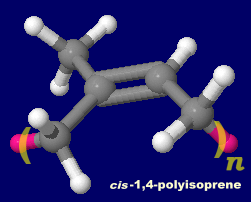
[右]6PPD-Q [左]ポリイソプレン(polyisoprene,cis-1,4結合;天然ゴムの主成分)
[Eastern equine encephalitis - Wikipedia]
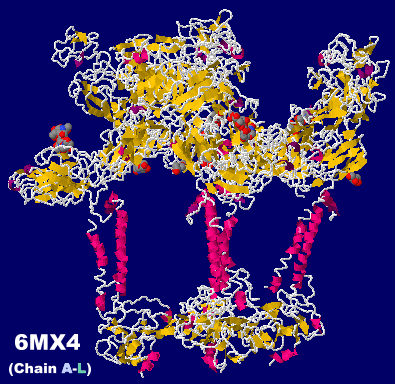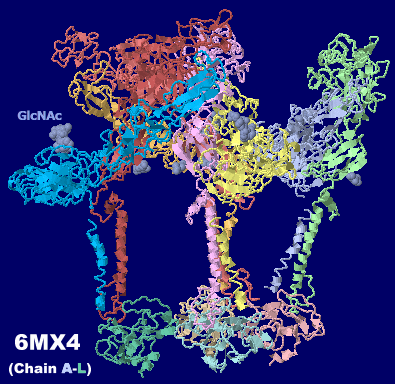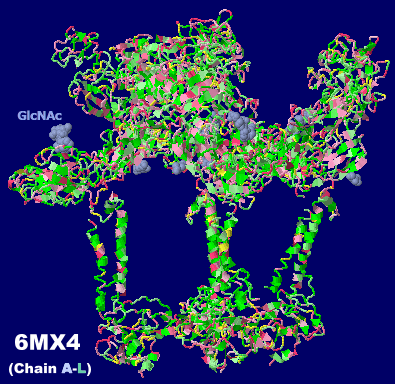
東部馬脳炎ウイルス(Eastern Equine Encephalitis Virus,EEEV)6mx4の部分構造(Chain A-L)
I = 170, O = 260, I/O = 0.654
[Dexmedetomidine - Wikipedia]
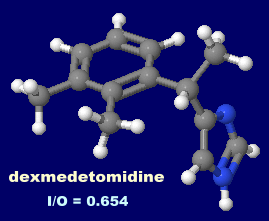
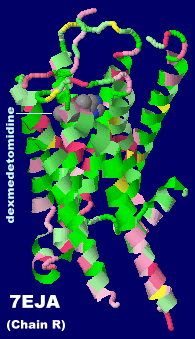
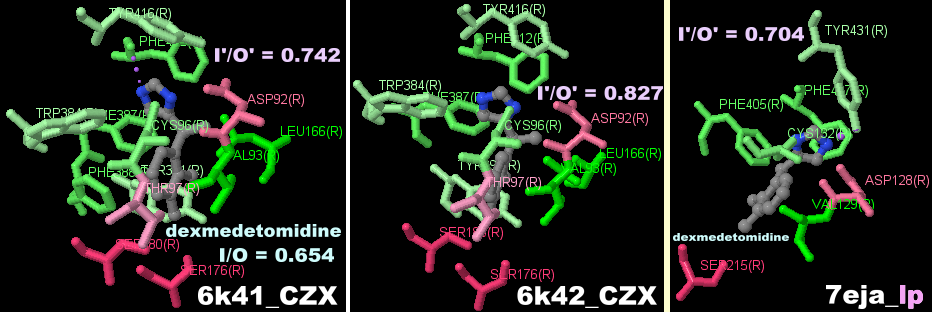
[左]デクスメデトミジン(dexmedetomidine)
[右]同分子結合アドレナリン受容体(α2AAR)7ejaとPDBsumデータ(6k41・6k42との比較)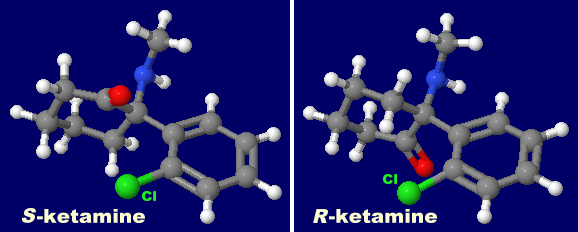
[左]S-ケタミン(S-ketamine)とR-ケタミン(R-ketamine)
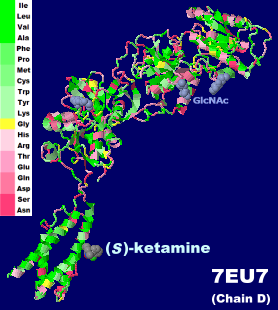
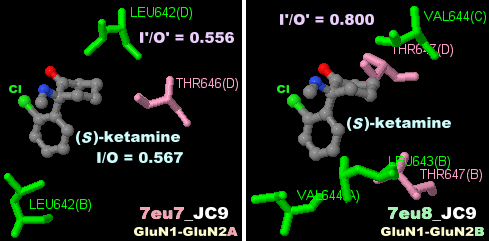
[右](S)-ケタミンが結合したGluN1-GluN2A NMDA受容体7eu7のChain DとPDBsumデータ(GluN1-GluN2Bの7eu8との比較)